R에서 GA Parameter에 따른 소요시간 테스트
지난 포스팅에 GA에 대한 기본 원리와 Objective function이 여러 개 일 때 GA로 최적화하는 방법에 대해 알아보았다.
[Optimization] 최적화 알고리즘 :: GA(Genetic Algorithm, 유전 알고리즘)란? GA 예시, R로 GA 구현하기
제조 공정에서 최적화란? 딥러닝을 공부한 사람이라면 최적화(Optimization)이라는 단어를 많이 들어보았을 것이다. 딥러닝에서 모델을 학습하는 과정에서 Cost function 값을 최소화 시키기 위한 Weight
leedakyeong.tistory.com
Multi-Objective Optimization(GA) :: Objective function이 여러개 일 때 Genetic Algorithm 원리, R code, 예시
이전 포스팅에서 Genetic Algorithm의 원리와 R에서 GA 라이브러리 사용법에 대해 알아보았다. 2022.01.12 - [AI/Optimization] - [Optimization] 최적화 알고리즘 :: GA(Genetic Algorithm, 유전 알고리즘)란? GA..
leedakyeong.tistory.com
[R] GA 특정 값으로 최적화 하는 방법 (no Maximizing) :: How to optimize with GA specific values in R
R에서 GA 최대값으로 최적화 하지 않고, 지정된 값으로 최적화 하는 방법 Genetic Algorithm(GA - 유전 알고리즘) 최적화 문제를 해결하는 기법 중 하나로, 생물의 진화를 모방한 방법이다. R에서는 GA 패
leedakyeong.tistory.com
이번에는 R에서 GA 파라미터별로 최적값을 찾기까지 소요되는 시간과 얼마나 최적값을 잘 찾아주는지 성능에 대해 알아보겠다.
공정에서 사용되는 전압을 최소화시킬 수 있는 최적의 공정 운전조건을 GA를 통해 찾는다 가정한다.
(여기서 Objective function은 공정 운전 조건을 X(변수)로, 전압을 Y(Target)으로 하는 예측 모델이다.)
GA의 가장 대표적인 파라미터인 popSize, maxiter, pcrossover, pmutation 별로 조금씩 바꿔보며 소요시간이 어떻게 달라지는지, 또 얼마나 최소값을 잘 찾아주는지 알아보겠다.
실험하고 있는 파라미터 외에는 모두 default값으로 고정하고 진행했다.
1. popSize
popSize는 각 세대별로 Simulation할 염색체 수 이다.
popSize를 제외한 다른 파라미터들은 default값으로 고정하고, popSize만 10, 20, ..., 100으로 바꾸어가며 확인했다.
(popSize의 Default값은 50이다.)
결과는 다음과 같다.
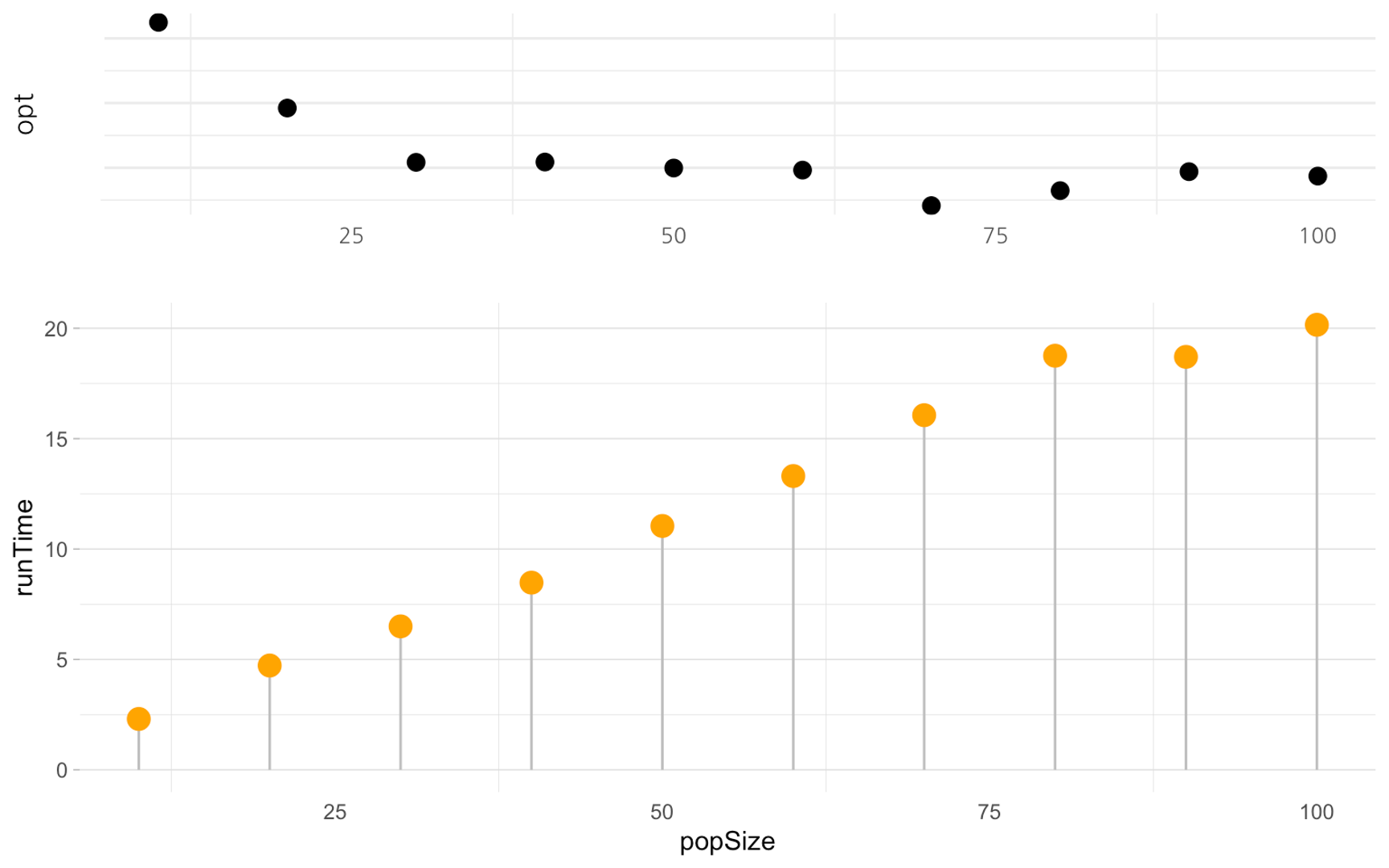
X축은 popSize를 10부터 20, 30, ..., 100까지 10씩 늘려본 값이고,
위 그래프의 Y축은 GA가 찾은 최적의 Target값, 아래 그래프의 Y축은 최적값을 찾기까지 소요된 시간(GA실행 종료시간-시작시간, 초)이다.
popSize가 커질수록 당연하게도 소요시간도 길어진다. 성능도 대체로 더 좋아진다.(최소값을 찾는 것이므로 값이 작아질수록 성능이 좋다고 판단한다.)
2. maxiter
maxiter는 iteration 돌릴 횟수이다.
R에서 Default값은 100이다.
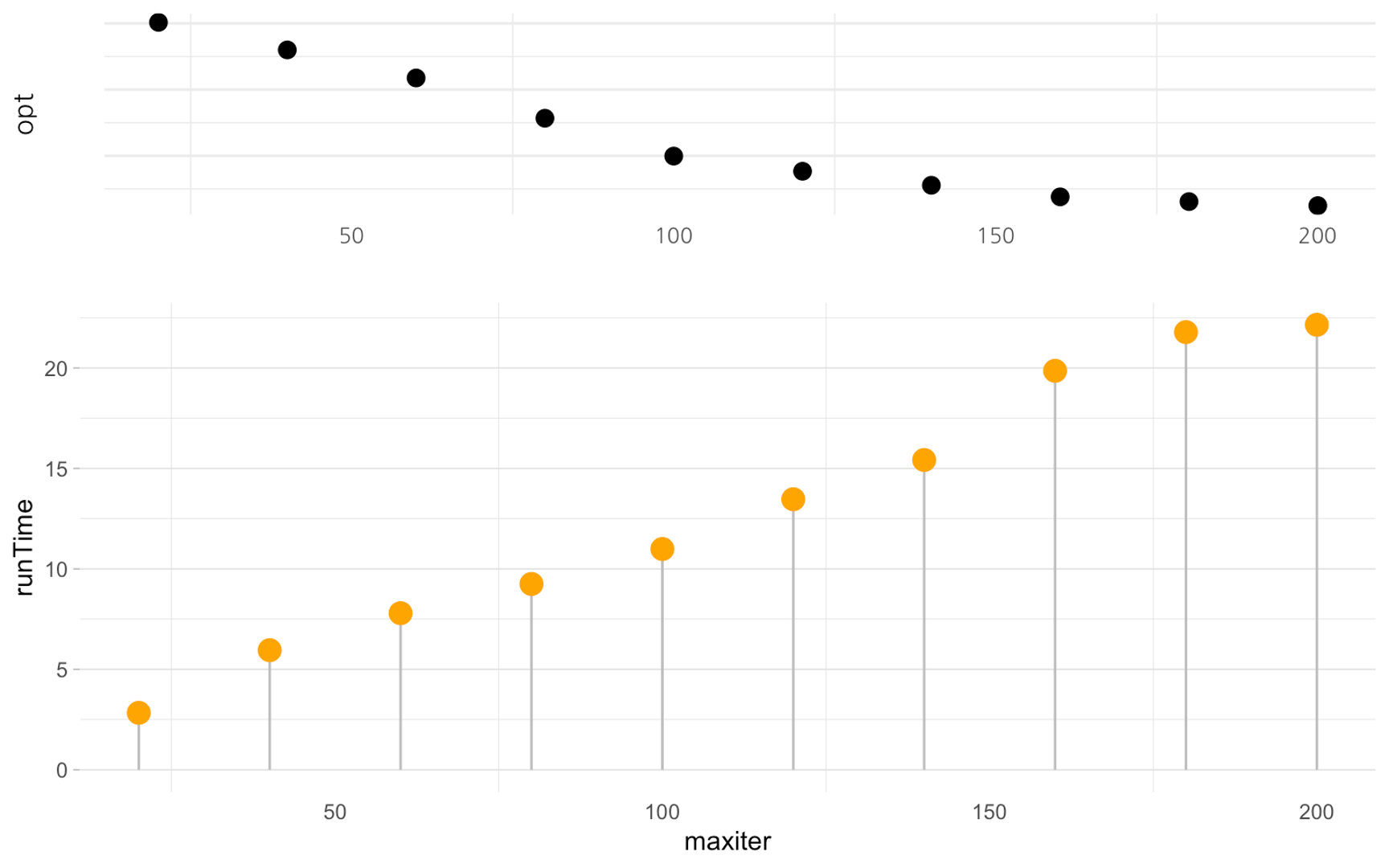
20부터 200까지 20씩 늘려가며 실행해본 결과이다.
역시 값이 커질수록 소요시간도 늘어나고 성능도 좋아진다.
3. pcrossover
pcrossover 교차를 진행할 염색체 수를 정하는 파라미터이며, Default값은 0.8이다. (0~1 사이값을 넣어주며, 확률을 의미한다.)

0.1부터 1까지 0.1간격으로 수행했다.
교차해야할 염색체수가 많아질수록 소요시간은 늘지만, 성능은 좀 좋아지다가, 오히려 안좋아지는 양상을 보인다.
4. pmutation
마지막으로 pmutation은 변이를 수행할 유전자 수를 정해주는 파라미터이다. Default값은 0.1이며, 역시 0~1 사이값을 전달해주어야 하고 percent를 의미한다.

변이는 많이 일으킨다고 해서 딱히 소요시간이 많이들지도, 성능이 좋아지거나 나빠지지도 않는다.
위 실험은 어떤 특정 problem에 대해 실험해 본 것이기 때문에 모든 문제에 적용되지는 않으나, 대략적으로 이렇다 라는 insight를 얻을 수 있었다.
popSize와 maxiter는 값이 커질수록 성능도 좋아지나, 소요시간도 길어지기때문에 실시간으로 대량의 혹은 복잡한 문제를 해결해야 한다면 타협하여 조절해야하며,
pcrossover와 pmutation은 문제에 따라 어떤 값이 좋을 지 많이 실험해보아야 할 것이다.